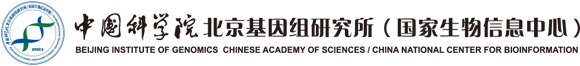北京基因組所(國家生物信息中心)薛勇彪研究員合作揭示古老轉座子擴張影響普通小麥轉錄調控演化
普通小麥是全球種植范圍最廣的谷物,具有廣泛的環境適應性,這被歸因于三套適應不同環境基因組的融合。基因序列相對保守,三套亞基因組的基因間區高度分化,那么,亞基因組如何實現調控的協同與分化呢?2022年11月14日,Nature Communications 在線發表了題為“Transposable elements orchestrate subgenome-convergent and -divergent transcription in common wheat”的研究論文,揭示了百萬年前轉座子的擴張影響普通小麥亞基因組轉錄調控的協同與分化。
本研究通過刻畫189個轉錄因子的全基因組結合模式(圖A),揭示小麥轉錄因子的結合位點在全基因組曾經發生多輪擴張(圖B)。進一步對調控圖譜開展進化分析,發現這些擴張與特定轉座子(TE)家族的擴張密切相關。因而亞基因組特異TE家族擴張貢獻于亞基因組的調控分化(圖C)。
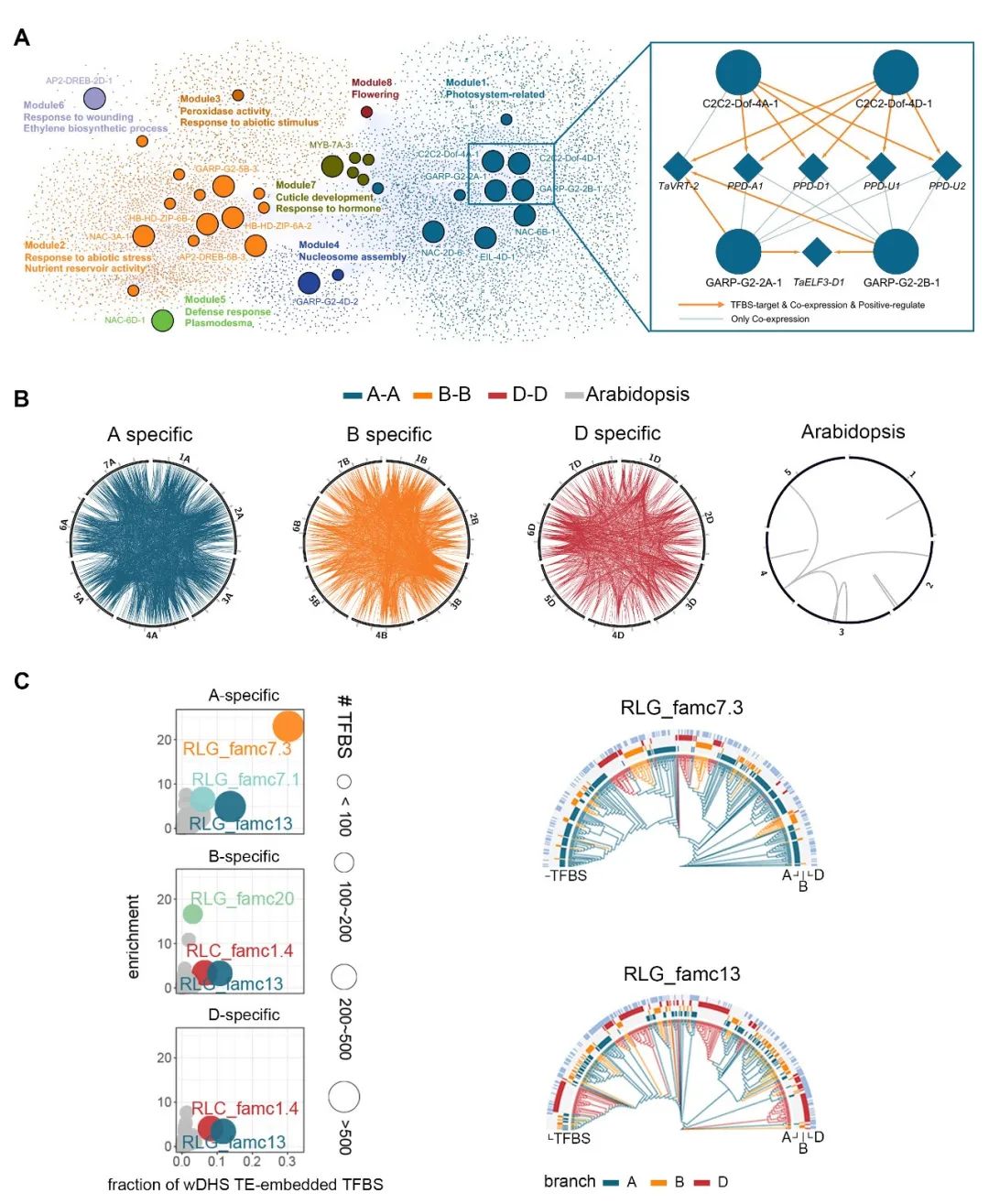
但是,高度分化的基因間區如何介導亞基因組協同調控呢?科研人員發現亞基因組協同的轉錄因子結合位點,大多只在一個亞基因組中由TE貢獻(圖D)。這樣不平衡的TE插入為何伴隨平衡的轉錄因子結合?研究人員推測,小麥基因組曾發生活躍的TE爆發和蛻化,導致很多TE不再具有經典的TE結構。通過設計統計檢驗策略,在全基因組水平鑒定基因附近蛻化的TE,發現三個亞基因組經歷了不平衡的TE蛻化(asymmetric TE decay),而內部轉錄因子結合位點在亞基因組之間高度保守,受到平行選擇(parallel evolution of TFBS)(圖E)。追溯這些TE的類型和爆發時間,發現亞基因共同祖先中一個特定TE家族(RLG_famc1.4)的古老擴張(約500萬年前),貢獻于普通小麥大量轉錄因子的協同調控(圖F)。結合轉錄組數據,發現這些轉錄因子平衡結合的靶基因在亞基因組間趨向于平衡表達。
綜上,該研究通過整合普通小麥轉錄因子結合圖譜并開發降解TE的檢測方案,發現500萬年前古老的TE擴張和亞基因組分化后新興的TE擴張對普通小麥亞基因組協同和分化調控的不同貢獻,展示了轉座子池的可塑性如何影響多倍體調控的可塑性(圖G)。
復旦大學張一婧研究員、中國科學院北京基因組所(國家生物信息中心)/中國科學院遺傳與發育生物學研究所薛勇彪研究員、中國科學院分子植物科學卓越創新中心/南方科技大學郎曌博研究員為論文的共同通訊作者。中國科學院分子植物科學卓越創新中心博士生張郁蕓、李子娟博士、博士生劉津易、中國科學院遺傳與發育生物學研究所張玉娥副研究員為論文的共同第一作者。南京農業大學張文利教授和中國科學院遺傳與發育生物學研究所童依平研究員合作參與本項工作。本項目得到中國科學院戰略先導項目,國家自然科學基金優秀青年科學基金項目,以及創新研究群體項目的資助。