北京基因組所(國家生物信息中心)開發基于低測序深度ATAC-seq數據的核小體排布和染色質開放性檢測技術
核小體排布和染色質開放性是重要的表觀遺傳學信號,與基因表達調控,胚胎發育,組織分化等眾多生理過程間存在緊密聯系。目前,ATAC-seq因其對樣品要求低,處理簡單,可獲得多維度染色質狀態信息等優勢成為表觀遺傳學的明星技術之一。然而在稀有樣本或者單細胞樣本中,ATAC-seq數據仍然相對稀疏。現有生物信息學技術在稀疏的ATAC-seq數據中普遍靈敏度不足。因此,研發能適用于低測序深度ATAC-seq文庫的高靈敏度核小體排布和染色質開放性檢測算法是一個尚未解決的領域內難題。
近日,中國科學院北京基因組研究所(國家生物信息中心)張治華研究組以“DeNOPA: decoding nucleosome positions sensitively with sparse ATAC-seq data”為題在國際生物信息學領域核心期刊Briefings in Bioinformatics上報告了一種基于低測序深度ATAC-seq文庫的核小體排布和染色質開放性檢測技——deNOPA。不同于現有算法根據測序片段長度,將ATAC-seq文庫拆分為分別用于染色質開放性評估和核小體排布檢測的子文庫的思路,該研究發現,來自不同長度測序片段的Tn5酶切位點,在基因組上分布相似。于是,利用這一相似性,研究人員開發了deNOPA。
該方法創新性的將核小體檢測任務的核心問題,由尋找核小體中心轉換為尋找核小體連接區域,從而使所有測序片段均在核小體檢測任務中得到應用。通過對一系列來自不同物種、不同測序深度ATAC-seq文庫,包括單細胞ATAC-seq文庫的性能測試。該算法的核小體檢測靈敏度相比現有算法大幅度提升,而代價僅是核小體定位準確性的可接受損失。基于該算法給出的核小體位置進行的單細胞分群研究也獲得了比現有策略更高的分群精度。
最后,該研究通過對熱刺激狀態和正常狀態K562細胞核小體排布和染色質狀態的測定和對比,描述了哺乳動物細胞熱刺激反應中染色質狀態和核小體排布的變化。不同于酵母,K562細胞熱刺激反應中轉錄起始位點附近核小體缺失區域的位置維持穩定,核小體占位率變化與所在元件活性是否受熱刺激影響有關,而與受影響的方向無關。
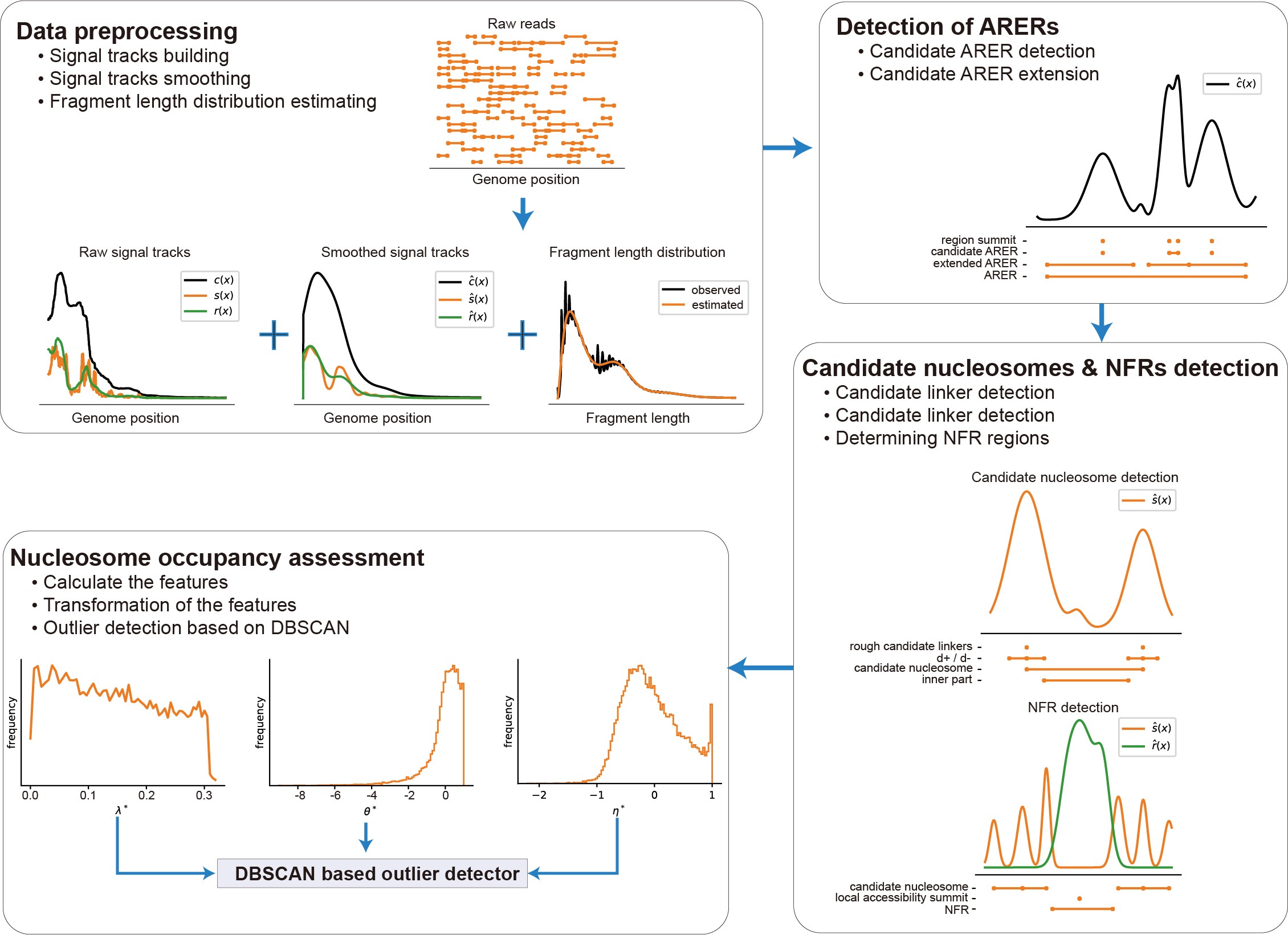
deNOPA算法的處理流程