北京基因組研究所(國家生物信息中心)合作發現沒有直接證據表明新型冠狀病毒RNA能夠整合進宿主基因組
新型冠狀病毒SARS-CoV-2是造成COVID-19在全球范圍肆虐的病因。該病毒為一種單股正鏈RNA病毒,具有高傳染性特性,能夠感染人體各代謝系統的多種組織器官,并引發嚴重的臨床癥狀,深入研究新冠病毒基因組RNA的特性十分必要。
2021年8月2日,中國科學院北京基因組研究所(國家生物信息中心)楊運桂研究組和中國醫學科學院醫學生物學研究所彭小忠研究組合作在Protein & Cell雜志上發表了題為“Comprehensive analysis of RNA-seq and whole genome sequencing data reveals no evidence for SARS-CoV-2 integrating into host genome”的研究成果。借助RNA測序(RNA-seq)和全基因組測序,研究團隊對SARS-CoV-2感染的人和猴細胞在轉錄組和基因組水平進行了分析,發現沒有直接證據表明新型冠狀病毒RNA能夠整合進宿主基因組。
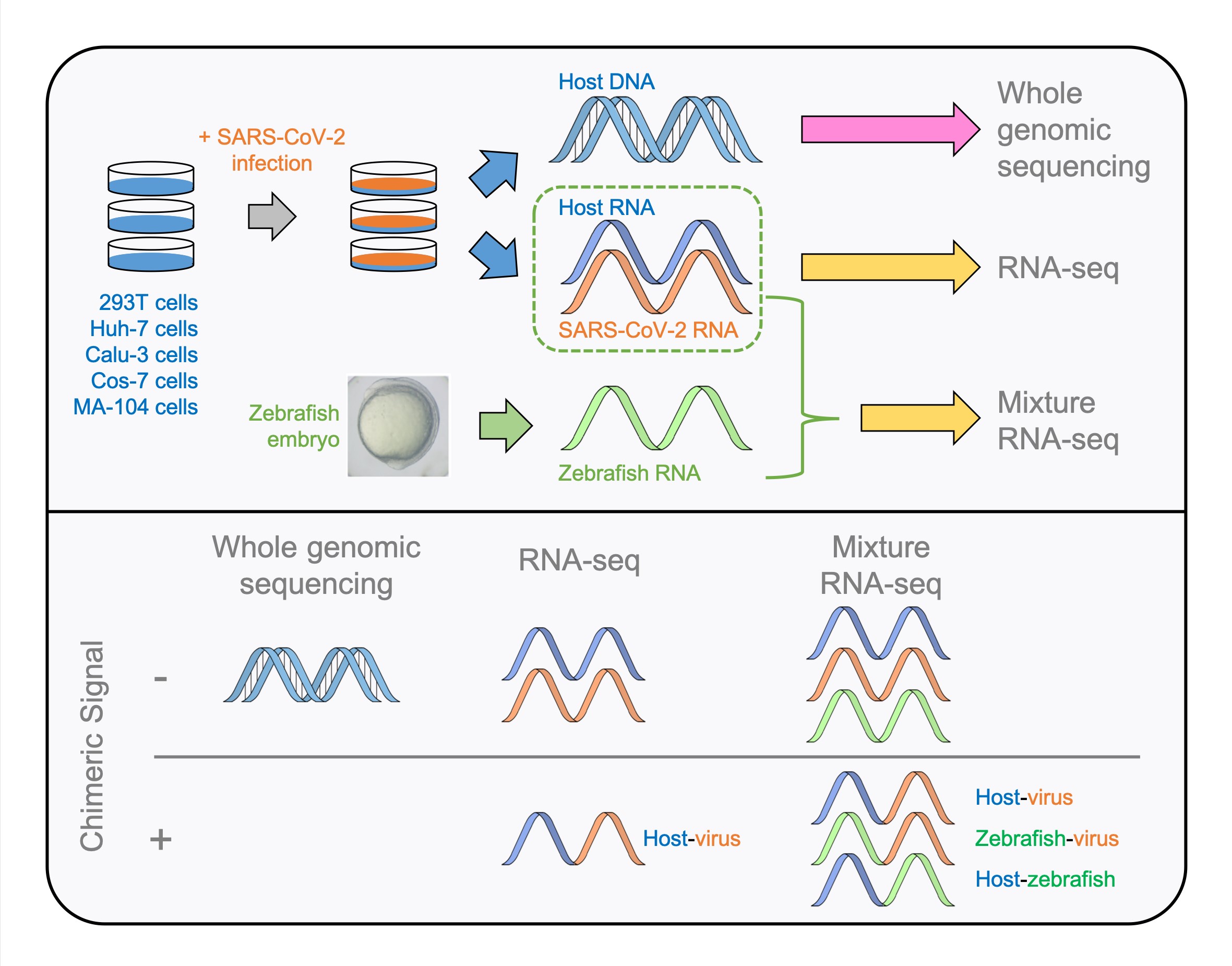
研究團隊首先用SARS-CoV-2分別感染了人類細胞系(293T、Huh-7和Calu-3)和猴細胞系(Cos-7和MA-104),并進行了轉錄組測序。分析發現,測序結果中存在宿主-病毒嵌合序列,其數量約占病毒測序讀段總量的0.5%-1.8%,但這些嵌合事件重復性極低,并與宿主基因表達水平正相關。之后對人類的三種細胞系進行全基因組測序,研究團隊并未在轉錄組高頻嵌合事件對應的DNA片段上鑒定到嵌合信號,甚至未檢測到任何能與SARS-CoV-2參考基因組匹配的DNA讀段。以上結果表明,SARS-CoV-2或并不具備整合進宿主基因組的能力,轉錄組測序數據中的嵌合序列可能來自其它途徑,如RNA測序文庫構建過程中的隨機連接。
為驗證該假設,研究團隊將SARS-CoV-2感染細胞和未感染斑馬魚胚胎的RNA進行混合,構建RNA測序文庫并進行轉錄組測序及分析。結果表明,除了人-病毒嵌合RNA片段,自然條件下不可能存在的斑馬魚-病毒嵌合RNA片段也出現在了轉錄組測序結果中,并且兩物種的各自嵌合讀段數與其在混合樣本中RNA豐度比例幾乎一致,證明了嵌合RNA讀段來自文庫構建過程中的隨機連接的推論。本研究通過對轉錄組測序、全基因組測序以及混合樣本轉錄組測序結果的系統性分析,發現并沒有直接證據表明SARS-CoV-2能夠整合進宿主基因組,其嵌合信號主要來自于建庫過程的隨機連接。
該研究得到科技部國家重點研發計劃等基金資助。
轉錄組及全基因組測序揭示新型冠狀病毒RNA不具備整合進宿主基因組的能力






