北京基因組所(國家生物信息中心)建立基于誘導多能干細胞技術的單細胞突變圖譜解析策略
近日,中國科學院北京基因組研究所(國家生物信息中心)蔡軍研究組與王前飛研究組合作在學術期刊Computational and Structural Biotechnology Journal發表題為A promising iPS-based single-cell cloning strategy revealing signatures of somatic mutations in heterogeneous normal cells的論文,該研究通過設計細胞培養實驗和基因組比較分析方法,揭示了誘導多能干細胞(iPSCs)的單細胞起源和基因組變異的來源組成,構建了單細胞突變圖譜解析策略。
為了準確檢測低頻的體細胞突變,比如正常細胞群體中的體細胞突變,當前已經有研究開發了MDA、MALBAC、 LIANTI等單細胞直接測序技術,但由于單個細胞的DNA含量非常少,這些方法存在擴增不均一、等位基因丟失等問題。單細胞體外培養擴增技術可通過體外培養將單個細胞培養成細胞株然后進行測序,通過識別細胞株中細胞群體的高頻率突變來間接觀測單個細胞基因組的體細胞突變情況。該技術基因組覆蓋度高、擴增錯誤少,但只適用于個別類型的干細胞。基于此,結合實驗設計和基因組比較分析方法,研究人員解析了誘導多能干細胞基因組變異來源的組成,證明了誘導多能干細胞的單個體細胞起源,以及單個體細胞與所形成的誘導多能干細胞在全基因組變異圖譜上超過90%的相似性,并進一步提出了基于誘導多能干細胞技術實現細胞克隆擴增的單細胞基因組變異解析的新策略,該策略可用于解析體細胞的基因組變異和譜系演化關系。
中國科學院北京基因組研究所(國家生物信息中心)蔡軍研究員與王前飛研究員為本文的共同通訊作者。中國科學院北京基因組研究所(國家生物信息中心)苗雪霞博士和李玥瑩博士為共同第一作者。該研究得到了國家重點研發計劃、國家自然科學基金、中國科學院戰略性先導科技專項等基金資助。
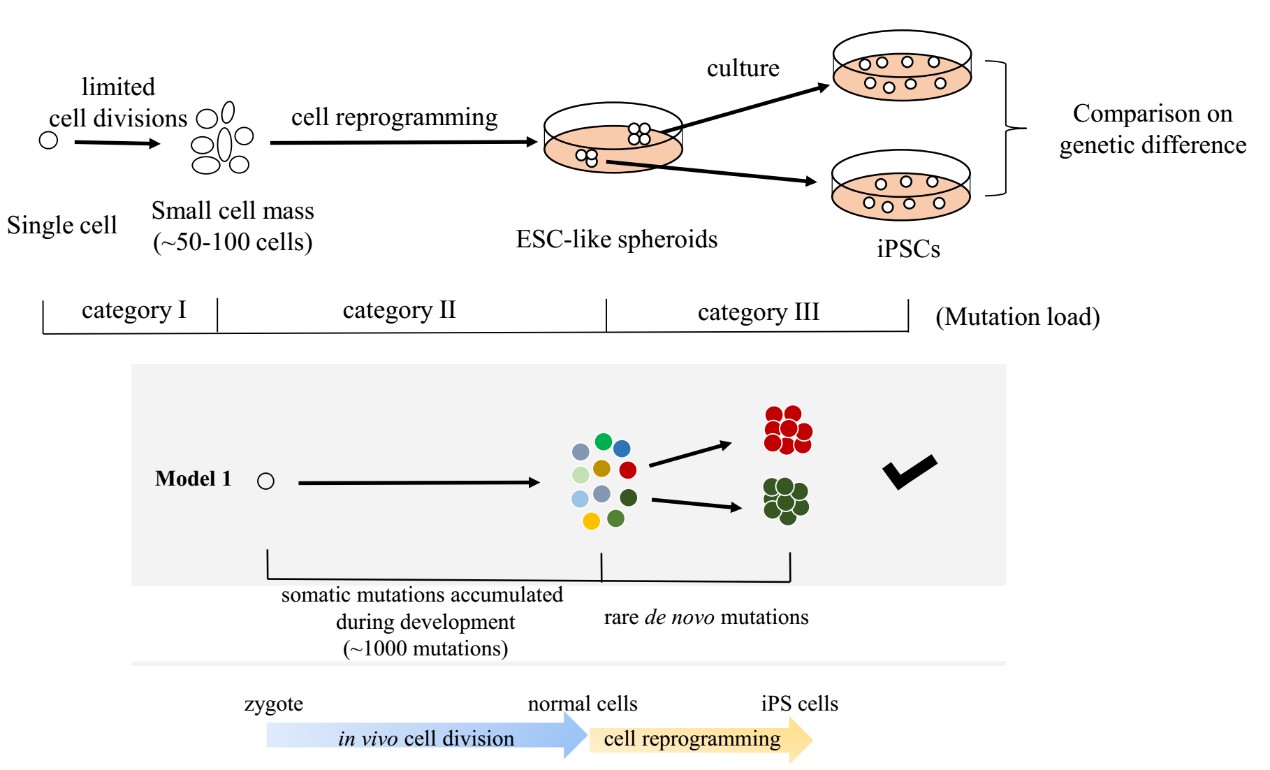
誘導多能干細胞基因組變異來源組成的解析策略和克隆擴增模型






