北京基因組所開發基因組變異-表型關聯知識庫
近日,由基因組所開發的國際上首個動植物基因組變異-表型關聯知識庫(GWAS Atlas)正式上線發表,該項研究成果以“GWAS Atlas: a curated resource of genome-wide variant-trait associations in plants and animals”為題在國際學術期刊《核酸研究》(Nucleic Acids Research)在線發表。
全基因組關聯分析(GWAS),即指在全基因組范圍內找出存在的序列變異,從中篩選出與疾病、表型性狀等相關聯的遺傳位點,是挖掘生物復雜性狀遺傳基礎的關鍵技術。隨著高通量測序技術的快速發展,多個物種產生了越來越多的高質量基因型數據,并已解析了與許多動植物復雜性狀相關聯的遺傳位點。但這些知識信息都分散在不同文獻中,不利于知識整合、挖掘與再利用。為此國家基因組科學數據中心的科研人員通過關鍵詞檢索、人工審編、詞條比對注釋等技術手段,結構化整理了現有主要農作物和畜牧動物的基因型-表型關聯知識,開發了世界上首個動植物基因組變異-表型關聯知識庫GWAS Atlas。
GWAS Atlas知識庫整合了9個物種(包括棉花、梅花、玉米、油菜籽、水稻、高粱、大豆7種植物和山羊、豬兩種動物)與614個性狀關聯的75467條基因型-表型(G2P)信息,并通過語義比對等映射到五個不同的性狀本體上(植物性狀本體PTO,家畜性狀本體ATOL,作物本體CO等),方便用戶通過基于本體的層級結構來查找感興趣的性狀及對應的G2P關聯信息。此外,研究人員還分析并定義了與多個性狀相關聯的多效基因及遺傳位點,支持用戶通過不同模塊在線瀏覽、檢索與下載。
GWAS Atlas作為首個綜合多物種的變異-性狀關聯知識庫,是全基因組變異信息數據庫(GVM)的延伸,將為未來重要農藝性狀的模塊化遺傳研究和育種應用提供重要資源和平臺。該研究得到了中國科學院戰略性先導科技專項、中國科學院國際大科學計劃等項目基金的資助。
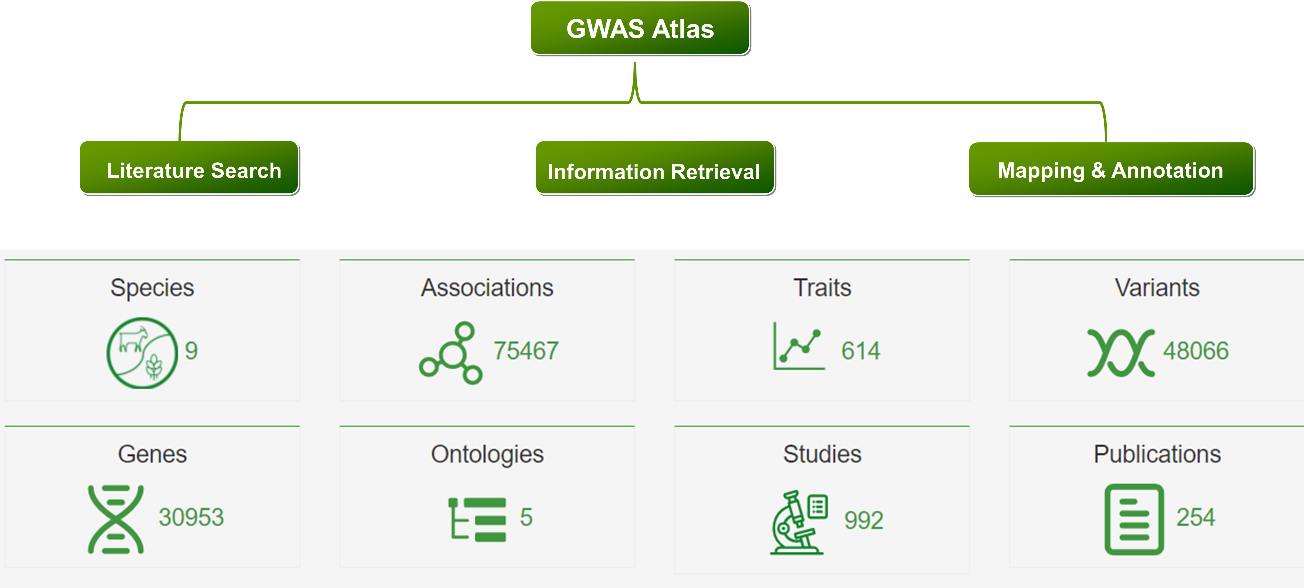
GWAS Atlas 資源信息統計






