北京基因組所合作開發比較基因組學進化分析新方法
比較基因組學是從進化角度分析不同物種的基因組數據,解析基因功能和疾病、表型的遺傳學機制。通過同源基因編碼區序列的進化比較是其中最常見的分析方法之一,如PAML等方法,都在物種序列比較分析中被廣泛應用。但這些方法僅分析多個物種的單一序列和分歧位點信息。隨著二代、三代測序技術的發展,眾多物種的基因組測序都已完成,越來越多的物種都在種內水平有了多個樣本的群體基因組數據。如果能將多物種群體水平的遺傳多態和物種水平的進化相結合進行分析,將有助于解析物種(尤其是近緣種)產生過程中適應性進化和特有表型形成的機制。迄今為止,尚且缺乏此類方法。
中科院北京基因組所陳華研究組與昆明動物所合作,首次開發了能夠同時分析多個物種的群體基因組數據的方法HDMKPRF。該方法以Hartl、Bustamante等的泊松隨機場模型和McDonald-Kreitman檢驗為框架,通過多個物種的聯合等位基因頻譜理論構建群體遺傳學模型,有效整合了微進化過程與宏觀進化。與現有分析方法比較,該方法采用貝葉斯方法,很大程度提高了對自然選擇基因的檢測功效;通過多個物種的群體基因組比較分析,能夠有效把自然選擇發生時間定位在多物種進化樹的具體某個階段(分支)上。該方法還提供了對各個物種的群體大小、物種分化時間以及自然選擇強度等參數的后驗概率分布。
利用該方法,研究人員對現代人、黑猩猩、大猩猩和猩猩四個靈長類物種基因組數據做了分析,在各個物種中鑒定了受到自然選擇而快速進化的基因。發現在人的特異性進化中,有84個與表達調控相關的基因受到正選擇,廣泛分布在鋅指蛋白基因家族、小RNA調節通路、TP調控、組蛋白修飾基因等不同類型中,印證了進化學家Allan Wilson和Mary-Claire King在1975年提出的觀點,即基因調控是人與黑猩猩在99%基因組序列相似基礎上表型高度差異的主要因素之一;發現與免疫、代謝等相關的通路受到了正選擇。此外,有大量的現代人與其他大猿分歧的基因富集在精神分裂癥、神經系統疾病相關的通路上,部分快速進化基因集中在精子生成、生殖相關通路上。該分析提供了靈長類四類大猿基因進化在時間軸和基因組上的自然選擇圖譜,為后續的進一步功能解析研究提供了基礎。
該研究于3月6日在線發表在進化生物學期刊《分子生物學與進化》上。研究得到了國家自然科學基金委重大研究計劃“微進化過程的多基因作用機制”、中科院院先導專項“動物復雜性狀的進化解析與調控”等項目支持。
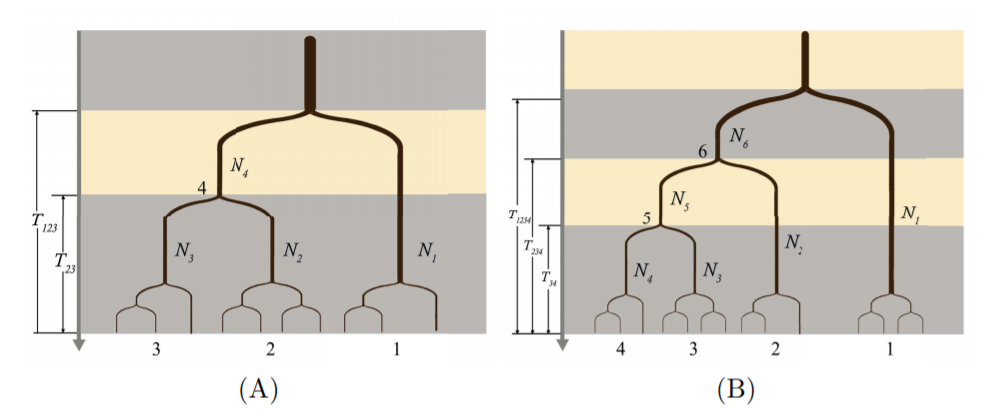
HDMKPRF方法的模型和和參數示意圖
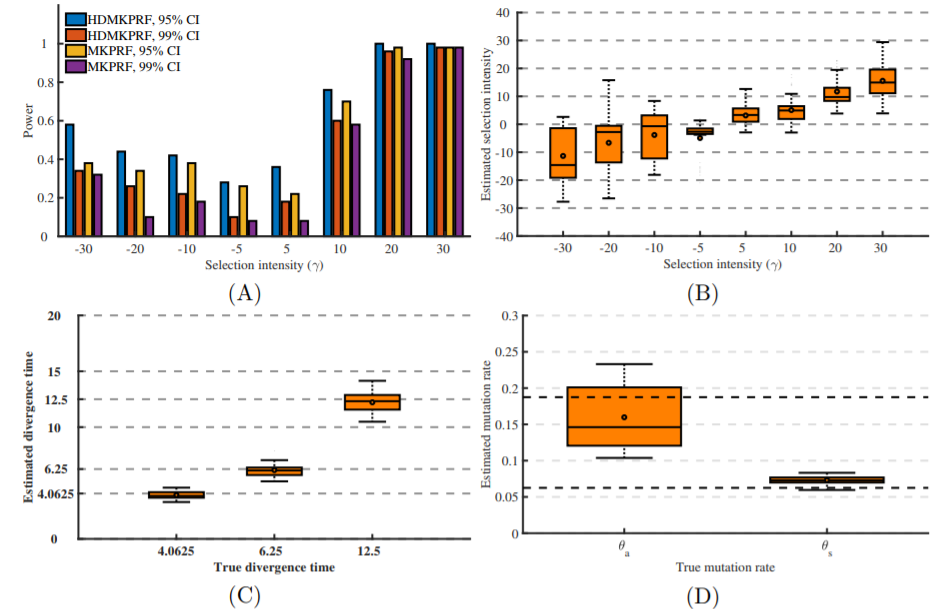
應用電腦仿真對HDMKPRF在檢測功效、對分歧時間、選擇強度等參數推斷上進行性能檢驗