北京基因組所開發跨物種長非編碼RNA鑒定軟件LGC
近日,基因組所在國際生物多樣性與健康大數據(Global Biodiversity & Health Big Data,簡稱BHBD)聯盟框架下,與沙特阿卜杜拉國王科技大學、巴基斯坦真納大學以及美國梅奧醫學院開展科研合作,聯合開發了可跨物種使用的長非編碼RNA(long non-coding RNA, lncRNA)鑒定軟件LGC。該成果以“Characterization and identification of long non-coding RNAs based on feature relationship”為題在國際學術期刊Bioinformatics在線發表。
測序技術的飛速發展產生了大量轉錄組數據,而轉錄組分析中一個重要的研究內容是鑒定新的轉錄本并且檢測其蛋白編碼能力。雖然利用蛋白質譜檢測、Ribo-seq等實驗方法可以獲得蛋白編碼證據,但成本高、耗時長且通量低。因此,目前主要依賴軟件預測的計算方法鑒定lncRNA。已有相關軟件大多需要高質量的訓練集,對于缺乏高質量數據的非模式物種,鑒定其lncRNA難度極大,亟需發展支持跨物種使用的lncRNA準確鑒定方法與工具。
為解決以上難題,北京基因組所聯合多家科研機構,研究了蛋白編碼RNA與lncRNA在多物種中普遍存在的序列特征差異,發現GC含量與ORF(開放讀碼框)長度的關系在兩者間存在顯著差異,且這種差異在動植物中廣泛存在。基于此,研發了能夠跨物種使用的lncRNA鑒定方法和軟件LGC。該方法不需要物種特異訓練集,與同類方法相比,LGC可實現從植物到哺乳動物的lncRNA的準確鑒定,且具有較為平衡的靈敏度與特異性,為lncRNA跨物種鑒定以及功能分析提供重要方法和工具。同時,提供web server,支持fasta、gtf、bed三種格式,實現在線分析計算。Fasta格式不限定物種,gtf與bed格式目前支持人、小鼠、斑馬魚與果蠅的在線分析。
該項研究得到了中國科學院戰略性先導科技專項、國家重點研發計劃、中國科學院“十三五”信息化專項等基金的資助。
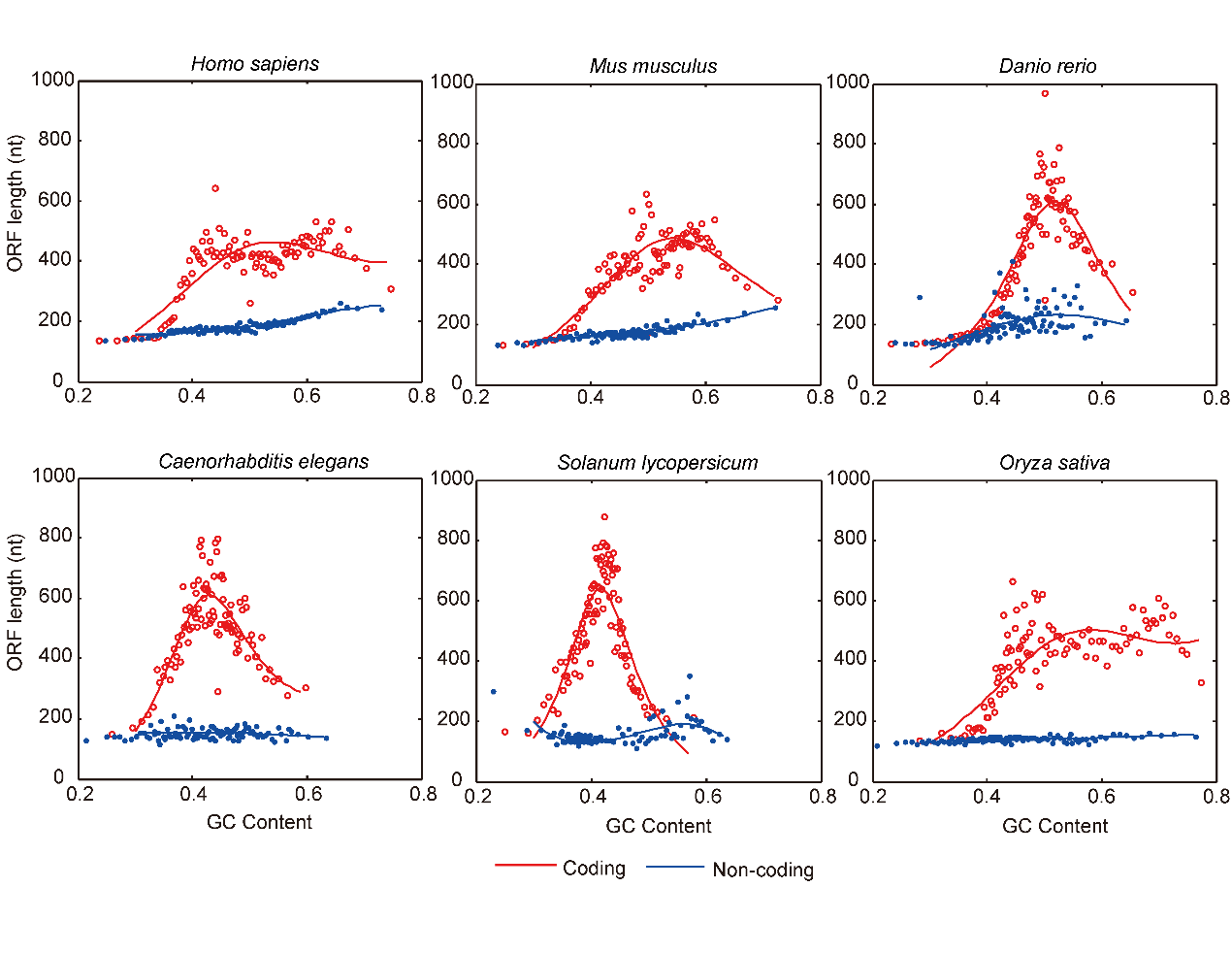
GC含量與ORF長度的關系在蛋白編碼RNA與lncRNA中存在顯著差異