北京基因組所合作開發單細胞測序數據中細胞子群特異DNA甲基化鑒定新方法
中國科學院北京基因組所呂雪梅研究組與美國弗吉尼亞理工大學謝荷煌研究團隊合作,利用小鼠胚胎干細胞得到的單細胞DNA甲基化測序數據,開發了一套生物信息分析流程,挖掘ESCs中的細胞子群特異DNA甲基化(CSM)。相關成果以“Integrative single-cell omics analyses reveal epigenetic heterogeneity in mouse embryonic stem cells”為題在線發表于PLoS computational biology雜志。
由于當前單細胞甲基化組數據具有覆蓋細胞數少、測序乘數低以及DNA片段擴增偏好等缺陷,導致對等位基因特異甲基化(ASM)、非對稱性甲基化(AM)在內的細胞內DNA甲基化異質性,和真正的CSM之間的區分能力十分受限。因此科研人員在過濾掉這些細胞內甲基化變異的干擾后,開發了新的統計模型,即“混合貝塔模型”以鑒定出真正的CSM位點。
該研究進一步整合了胚胎干細胞單細胞轉錄組數據、小鼠成體組織甲基化組數據和轉錄因子結合基序注釋信息,對CSM的調控模式進行解讀。結果發現CSM位點顯著富集到CGI shelf區域,以及增強子和啟動子偏好的組蛋白修飾區域;同時CSM定位到的基因在單細胞間表現出廣泛的表達差異。進一步研究發現,CSM位點可以被聚類成共甲基化的模塊,這些CSM模塊在不同成體組織中具有不同的甲基化水平分布,并且表現出模塊特異的轉錄因子富集。該方法為單細胞甲基化組測序數據表觀異質性的深度挖掘提供了方法學的創新,對于研究復雜細胞群體異質性具有重要指導意義。
該研究得到國家自然科學基金、中國科學院戰略性先導科技專項等基金及動物進化與遺傳前沿交叉卓越中心的資助。
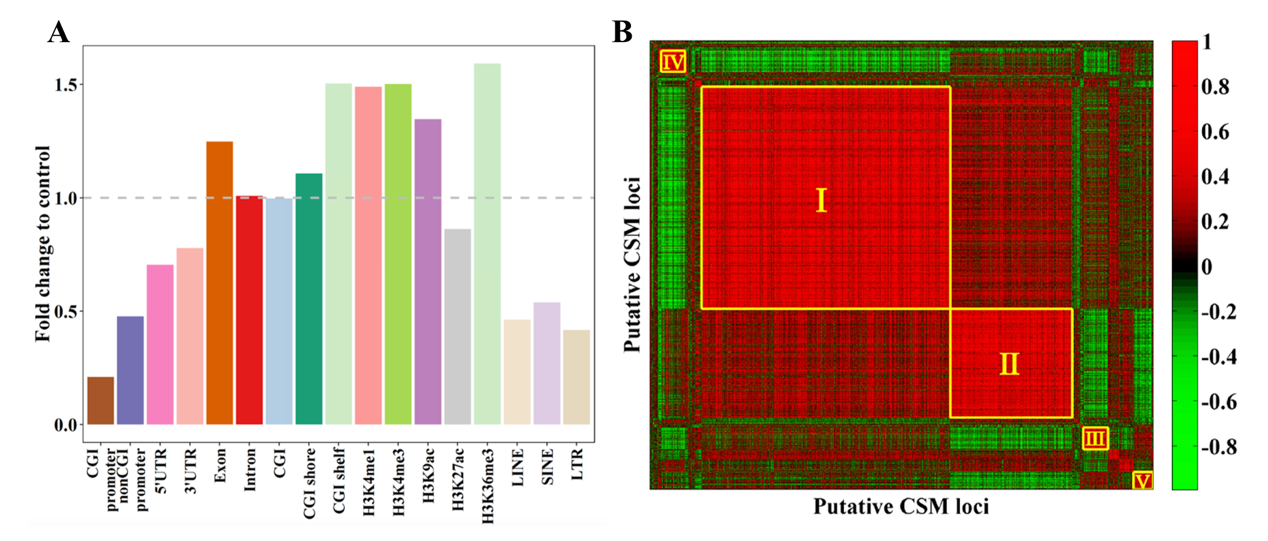
(A) CSM位點在不同基因組特征區域上的富集分布;(B) 兩兩CSM位點的皮爾森相關系數熱圖,前五
共甲基化CSM模塊按羅馬數字序標注;顏色從綠到紅表示相關系數從-1到1






