GSA:組學原始數據庫系統
生命科學的發展已進入組學大數據時代,然而中國至今尚未形成可服務于科學研究的公共數據庫存儲體系。為了彌補這一空白,中國科學院北京基因組研究所生命與健康大數據中心開發并構建了組學原始數據存儲歸檔系統Genome Sequence Archive(簡稱GSA;http://bigd.big.ac.cn/gsa 或 http://gsa.big.ac.cn)。GSA的系統建設遵循了國際核酸序列共享聯盟(International Nucleotide Sequence Database Collaboration, INSDC)的相關標準,并作為INSDC的補充,旨在減輕國際相關數據庫數據存貯及數據傳輸的壓力;立足中國,服務全球。
引言
第二代高通量測序技術革新推動了生命科學研究的縱深發展與應用,尤其在人口與健康領域,世界眾多國家相繼啟動了大型研究計劃,如美國的精準醫學研究計劃 [1]、英國萬人基因組計劃 [2]、冰島人群基因組計劃 [3]、中國精準醫學研究計劃 [4]等。這些研究計劃都將產生大量的組學數據,從而導致了生命健康組學大數據的爆炸性增長。與此同時,數據存儲、整合與挖掘、轉化與應用將成為重要的技術問題與挑戰 [5,6]。
國際上,美國、歐洲和日本于2005年建立了國際核酸序列共享聯盟(INSDC)[7],包括NCBI [8]、EBI [9]和DDBJ [10]三大數據庫系統,形成領域內數據存儲和共享使用的標準,接收并存儲來自全世界科學家提交的組學數據。然而,中國是一個生物資源大國,也是一個數據產出大國;迫于學術論文的發表及學術期刊的要求,中國的科學家需要將大量的數據跨過海底線纜,提交到國際數據庫。但由于中國國際網絡出口帶寬的瓶頸問題,數據傳輸效率低下。以中國科學院北京基因組研究所的150Mbs出口帶寬為例,向NCBI數據庫遞交1TB的數據需要花費2周以上的時間。當前,中國已經啟動國家級的精準醫學研究計劃以及若干大型的具有地域特色的研究任務。可以預見,未來中國每年將產生數十PB的組學數據;這將為目前的數據傳輸、存儲與共享提出新的挑戰。
為了緩解上述困難和問題,中國科學院北京基因組研究所開發并構建了組學原始數據庫系統GSA,專注于組學原始數據收集與整合,并提供免費的數據存儲、共享與訪問服務 [11]。GSA遵循國際INSDC的數據標準及數據庫建設標準,可收集來自不同測序平臺產出的數據,并存儲序列數據及其對應的元數據信息,確保數據的完整性。GSA立足于中國,極大的方便了中國科學家的數據遞交;同時,服務于全球,為全世界的科研領域共享并貢獻數據。
數據庫內容和使用
數據結構與模型
為了確保與INSDC數據庫系統的兼容性,GSA遵循了INSDC數據庫系統的數據標準和數據結構,并將數據分為四類,即項目信息(BioProject)、樣本信息(BioSample)、實驗信息(Experiment)和測序信息(Run);數據結構如圖1所示。
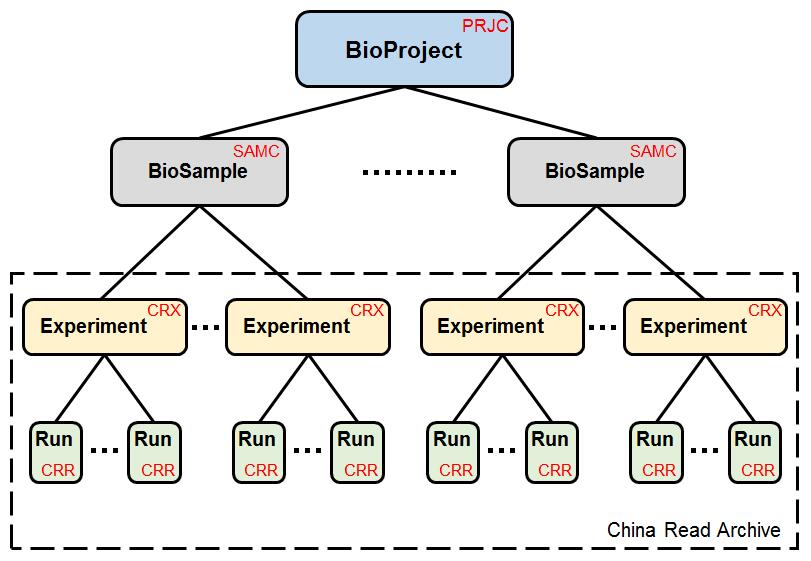
圖1 GSA數據模型
項目信息的數據獲取號(Accession Number)以“PRJCA”為前綴,其中字母“C”表示中國。項目信息提供了一個針對本研究任務的概要性描述,并包括研究目的、涉及的物種、數據類型、數據遞交者、基金資助機構、發表的文章等信息。樣本信息的數據獲取號以“SAMC”為前綴,包含一些有關生物樣本的描述信息如樣本類型、樣本屬性等。實驗信息以“CRX”為前綴,為特定樣本實驗處理方式,包括實驗目的、文庫構建方式、測序類型等信息。測序信息的數據獲取號以“CRR”為前綴,內容主要包括測序文件和對應的校驗信息。在四類數據中,項目信息和樣本信息是獨立運行的模塊,而實驗信息和測序信息形成了測序序列的歸檔庫。基于上述標準和結構,GSA不僅方便數據遞交,而且便于管理數據權限,實現數據共享與交換。
除此之外,GSA考慮大型項目管理的需求,引入Umbrella Project概念,提供大型合作型項目的傘裝結構管理。目前,已有兩個中國科學院戰略先導項目和一個中國科學院重點研究項目正在使用GSA系統管理和共享項目數據。
數據歸檔與統計
GSA接收來自全球的數據遞交,接收不同測序平臺產出的組學數據,并支持通用的數據文件格式如FASTQ、BAM、VCF。同時,GSA對接收到的數據進行質量評估,確保數據的完整性和可用性。在數據安全方面,類似于INSDC數據庫系統,GSA允許數據遞交者設置其數據的訪問權限(公開訪問或受控限制);公開即意味著數據可被任何人訪問或下載使用,受控即其他人的訪問在一段時間內將被受到限制。在GSA系統后臺,可被公開訪問和受控訪問的數據存儲于不同的磁盤空間內,以確保數據的安全性。從2015年8月份GSA系統上線至今,系統中的數據呈現顯著增長的趨勢(圖2),截止到2016年底,GSA已經接收了來自39個研究機構160余位科研人員的用戶注冊信息,并收錄198個項目,8674個樣本,9263個實驗和10745個測序信息,涵蓋了超過80個物種的信息。
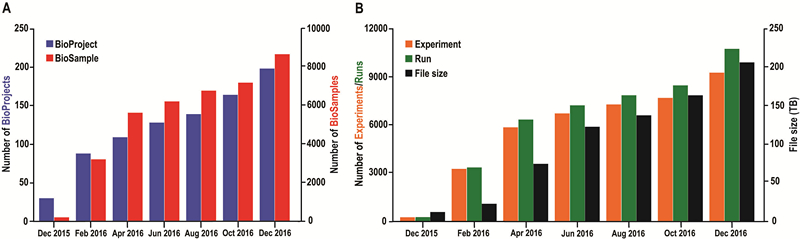
圖2 GSA數據統計
數據遞交與信息檢索
GSA系統提供用戶注冊和登錄功能,因此在創建一個數據遞交前,首先需要通過GSA系統注冊用戶賬戶,在用戶賬號被驗證通過并激活后,方可登錄系統并創建數據遞交頁面。通常情況下,在GSA中完成一個數據遞交需要執行五個操作,分別為注冊項目、樣本、實驗、測序四類元數據信息和提交序列文件(圖3)。在元數據信息收集頁面,GSA系統提供友好訪問的頁面向導幫助用戶實現信息錄入;而針對測序文件上傳,GSA提供基于IPV4和IPV6兩條網絡鏈路的FTP服務器,確保數據傳輸的高效性。GSA系統實現了數據全局檢索功能,并對檢索的結果進行分類統計;同時,用戶可以預覽檢索出的每一個數據的詳細信息。
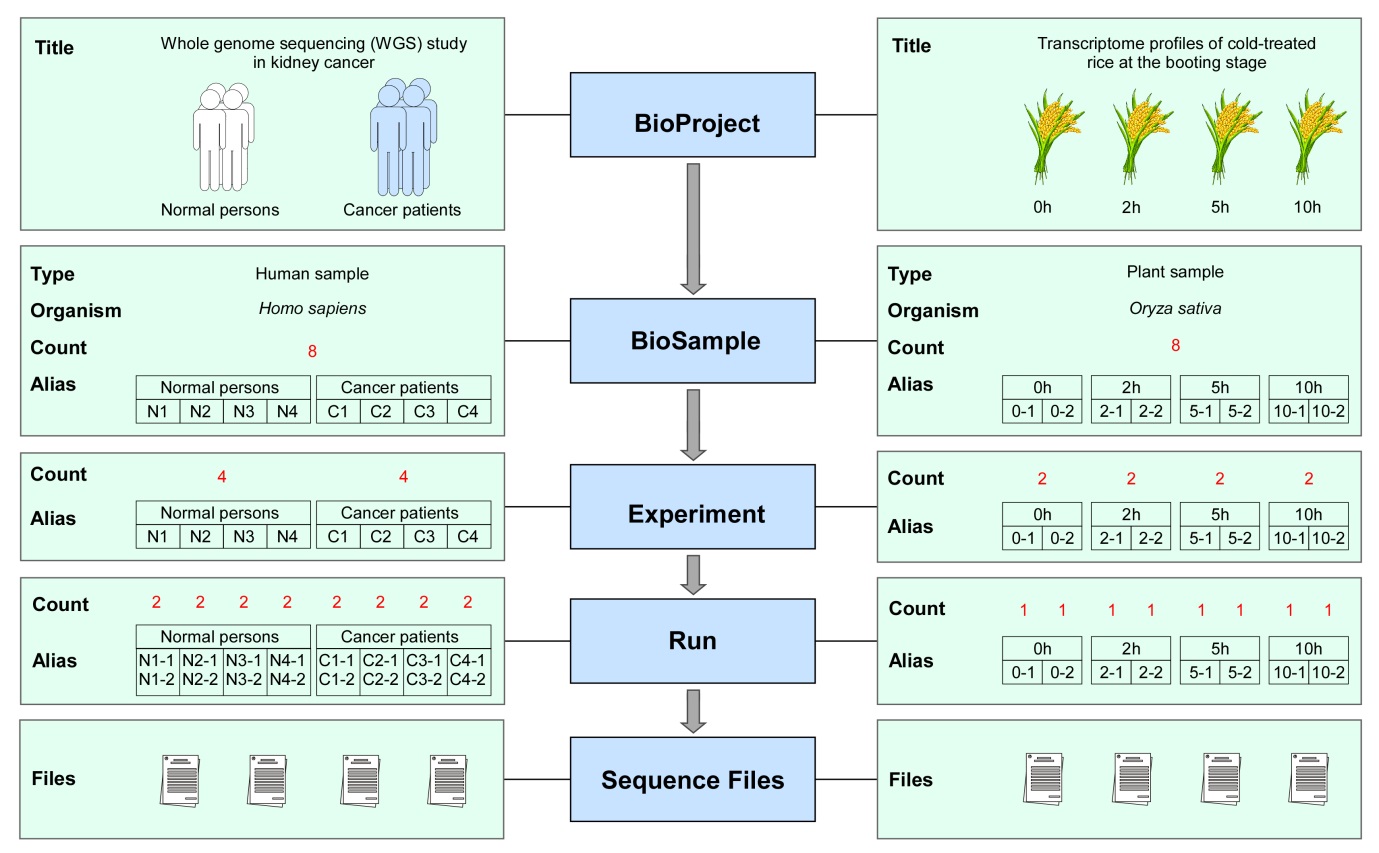
圖3 GSA數據提交
發展與展望
俗話說“能力有多大,責任就有多大”。當今的中國已是世界第一大經濟體,并在全球的經濟體中發揮著越來越重要的作用。同樣,在科研領域,在當今中國組學測序數據產量顯著增加的情況下,我們應該承擔起相應的責任,建立國際化的組學數據存儲體系,分擔國際數據庫數據存儲壓力,服務于全球的生命科學研究機構。
GSA與國際同類數據庫一樣,致力于存儲生命科學研究產出的組學大數據,并致力于中國組學數據匯交、管理、共享與應用體系的建設,促進中國在生命組學大數據領域的發展,提升中國在國際組學數據共享領域的地位,服務于全世界的生命科學研究與產業創新應用。 基于此,中國科學院北京基因組研究所發起“中國基因組數據共享倡議”(http://bigd.big.ac.cn/gdsd),呼吁中國產出的組學數據遞交GSA進行統一存儲、管理與共享。在倡議發出后很短的時間內,得到全國超過380個機構的1000余人支持本倡議。這代表了中國人的心聲,也代表了中國眾多科研資助機構的心聲。
總結
GSA是一個公共的、免費的組學原始數據存儲庫,在建設標準上遵循國際INSDC數據庫體系的數據標準和數據庫結構標準,在內容上收集生命科學研究中產生的組學測序數據及其元數據信息,并且接受來自全世界科研人員的數據遞交與獲取請求。在組學大數據時代,GSA不僅作為當前INSDC數據庫體系的補充以緩解組學大數據遠距離傳輸與儲存的壓力,而且承擔推動國際組學大數據共享的責任。
未來,GSA將逐步擴展與完善系統功能,提供專業化的組學大數據管理解決方案,如面向國家精準醫學研究計劃的組學大數據存儲與管理,面向宏基因組數據的存儲與管理等;另一方面將重點加強IT基礎設施的建設,并提升數據存儲能力和共享效率。
致謝
感謝羅靜初教授和朱偉民教授給予GSA系統建設的諸多寶貴意見和建議。本項目也得到了國家項目基金的支持,主要有:中國科學院先導項目、國家高技術研究發展計劃(863計劃)、國家重點研究發展計劃、中國科學院國際合作國際大科學計劃、中國科學院重點部署項目、中國科學院關鍵技術人才項目。
參考文獻
[1] Collins FS, Varmus H. A new initiative on precision medicine. N Engl J Med 2015;372:793–5.
[2] Taylor PN, Porcu E, Chew S, Campbell PJ, Traglia M, Brown SJ, et al. Whole-genome sequence-based analysis of thyroid function. Nat Commun 2015;6:5681.
[3] Gudbjartsson DF, Helgason H, Gudjonsson SA, Zink F, Oddson A, Gylfason A, et al. Large-scale whole-genome sequencing of the Icelandic population. Nat Genet 2015;47:435–44.
[4] Bai B, Zhao WM, Tang BX, Wang YQ, Wang L, Zhang Z, et al. DoGSD: the dog and wolf genome SNP database. Nucleic Acids Res 2015;43:D777–83.
[5] Xue Y, Lameijer EW, Ye K, Zhang K, Chang S, Wang X, et al. Precision medicine: what challenges are we facing? Genomics Proteomics Bioinformatics 2016;14:253–61.
[6] Zhang Z, Bajic VB, Yu J, Cheung KH, Townsend JP. Data integration in bioinformatics: current efforts and challenges. In: Mahdavi MA editor. Bioinformatics ––trends and methodologies. Rijeka: InTech;2011,p.41–56.
[7] Cochrane G, Karsch-Mizrachi I, Takagi T, International Nucleotide Sequence Database Collaboration. The International Nucleotide Sequence Database Collaboration. Nucleic Acids Res 2016;44:D48–50.
[8] NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Res 2016;44:D7–19.
[9] Cook CE, Bergman MT, Finn RD, Cochrane G, Birney E, Apweiler R. The European Bioinformatics Institute in 2016: data growth and integration. Nucleic Acids Res 2016;44:D20–6.
[10] Mashima J, Kodama Y, Kosuge T, Fujisawa T, Katayama T, Nagasaki H, et al. DNA data bank of Japan (DDBJ) progress report. Nucleic Acids Res 2016;44:D51–7.
[11] BIG Data Center Members. The BIG Data Center: from deposition to integration to translation. Nucleic Acids Res 2017;45:D18–24.
該文英文發表在Genomics, Proteomics & Bioinformatics期刊2017年第一期,全文鏈接http://www.sciencedirect.com/science/article/pii/S1672022917300025;http://gpb.big.ac.cn/






