北京基因組所對大樣本全基因組數據的群體遺傳學分析方法開發獲重要進展
計算基因組學旨在發展理論和方法學,對基因組數據進行數據挖掘、提取信息。其中對遺傳多態性,如基因頻譜、單倍型結構等進行建模,利用群體基因組水平的遺傳變異數據來推斷群體的歷史和變遷是群體遺傳學的核心內容。傳統的群體遺傳學分析方法通常基于小樣本數據,所推斷的多為相對古老的歷史事件,例如,被廣泛應用的Li and Durbin (2011)的PSMC方法(遞次式對偶溯祖方法)適用于2萬年到300萬年之間的群體大小變化,對一萬年以內群體歷史的推斷精度很有限。該時間區間是這些方法進行參數推斷的“盲區”。而人類在過去一萬年左右從漫長的狩獵-采集文明逐漸過渡到農業、畜牧業和工業文明,深入了解人類兩萬年內的群體進化和變遷,對解析環境適應性、遺傳性疾病的易感性和發病機制等都有重要意義。
日新月異的測序新技術正在產生海量的基因組序列數據。這些大樣本或群體水平的測序數據為基因組時代的群體遺傳學研究提供了前所未有的機遇。大數據蘊含的豐富信息使得更精細推斷群體歷史,包括1萬年乃至幾千年以內的群體變化成為可能。但另一方面,也給現有的理論和方法帶來了新的挑戰。多數現有的分析方法并不適用分析大數據:一方面是由于這些傳統方法大多是基于隨機取樣方法,計算量太大;另一方面則源于一些公式在大樣本條件下存在數值不穩定性。
中國科學院北京基因組研究所計算基因組中心陳華課題組針對以上問題提出了一個群體遺傳學新算法(TNSFS)。該方法克服了大樣本時的數值計算問題,首次實現了對大樣本全基因組數據進行計算高效的群體遺傳學分析,可用于檢測群體的增長模式,有效推斷一萬年以內的群體大小變化的相關參數。新算法擁有若干計算上的優勢:該方法給出解析形式的公式,不依賴于仿真,計算便捷高效,而且在大樣本時無數值問題;具有很好的靈活性,能涵蓋復雜的群體模型;此外,現代群體遺傳學模型以Kingman溯祖理論為基本構架,理論上只適用于樣本量遠小于群體大小的前提下。當樣本數目很大,甚至于接近群體水平時,Kingman溯祖理論會有嚴重偏差。新提出的方法即使在這種情況下,也具有很好的魯棒性(robustness)。
該工作的研究成果于2015年11月發表在進化生物學領域權威期刊 Molecular Biology and Evolution。
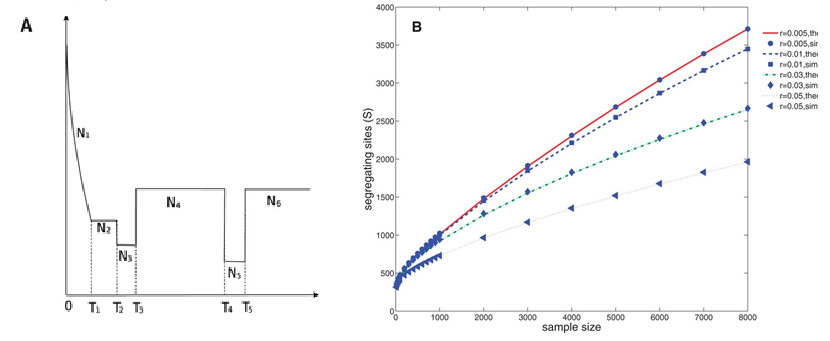
TNSFS方法能有效模擬復雜的群體模型(如圖A所示,用于描述歐洲群體變化的Gazave模型), 并且在不同參數值下的理論預測值與電腦仿真產生的結果吻合(圖B)






