基因組所合作開發完成基于K-mer的基因組組分分析數據庫
在過去的幾十年中,人們往往使用高度保守的基因家族進行系統進化分析,采用全基因組序列進行系統進化分析并不普遍。目前,基于是否進行序列的比對,分子系統發生樹的構建分為兩類。其中,不需要進行序列比對的方法是依據K-mer向量計算的距離矩陣進行系統進化分析,大量的研究證實該算法是行之有效的,尤其是對基因組中諸如蛋白編碼序列等的特定區域。不僅如此,K-mer算法還在組學的其他方面,包括基因組組裝、motif預測、重復序列的識別以及基因組的復雜性評估等都受到了廣泛的關注。基于K-mer算法在組學中的重要表現,在這個大規模基因組數據快速積累的時代,構建一個基于K-mer算法易于存儲并且將大量基因組數據可視化處理的數據庫十分迫切。
為此,中國科學院北京基因組研究所基因組科學與信息重點實驗室于軍組和英國倫敦大學學院(UCL)腫瘤研究所王大鵬博士合作開發了一套基于K-mer算法的基因組組分分析數據庫KGCAK。此項研究于近期發表在Biology Direct雜志。
在這個數據庫中,研究人員搜集了Ensembl、Phytozome和NCBI等幾大主流基因組數據庫中包括高等動植物、原生生物、真菌、細菌、病毒等在內的8000多個核基因組或者細胞器基因組,同時包括基因組不同維度的序列,主要有DNA、cDNA、CDS、氨基酸和ncRNA序列。并且還分別計算和存儲了核酸序列(K從2變化到10)和氨基酸序列(K從1變化到5)的K-mer向量,以方便進行不同維度數據跨物種的系統發生樹構建。此外,該數據庫提供了評估不同物種基因組復雜度的交互工具,主要包括基因組基本特征參數、K-mer向量的數學參數統計、頻率分布、唯一性比率,以及二維和三維空間可視化分析基因組參數和K-mer參數的交互關系等。
總的來說,該數據庫通過捕獲基因組序列特征并把基因組轉化成更易于理解和可視化的數字K-mer向量,以期通過K-mer算法用可視化的圖形和定量的數據構建一個比較基因組學的平臺,將為系統發生樹構建和通過基因組數據研究物種關系提供良好的參照和指引。
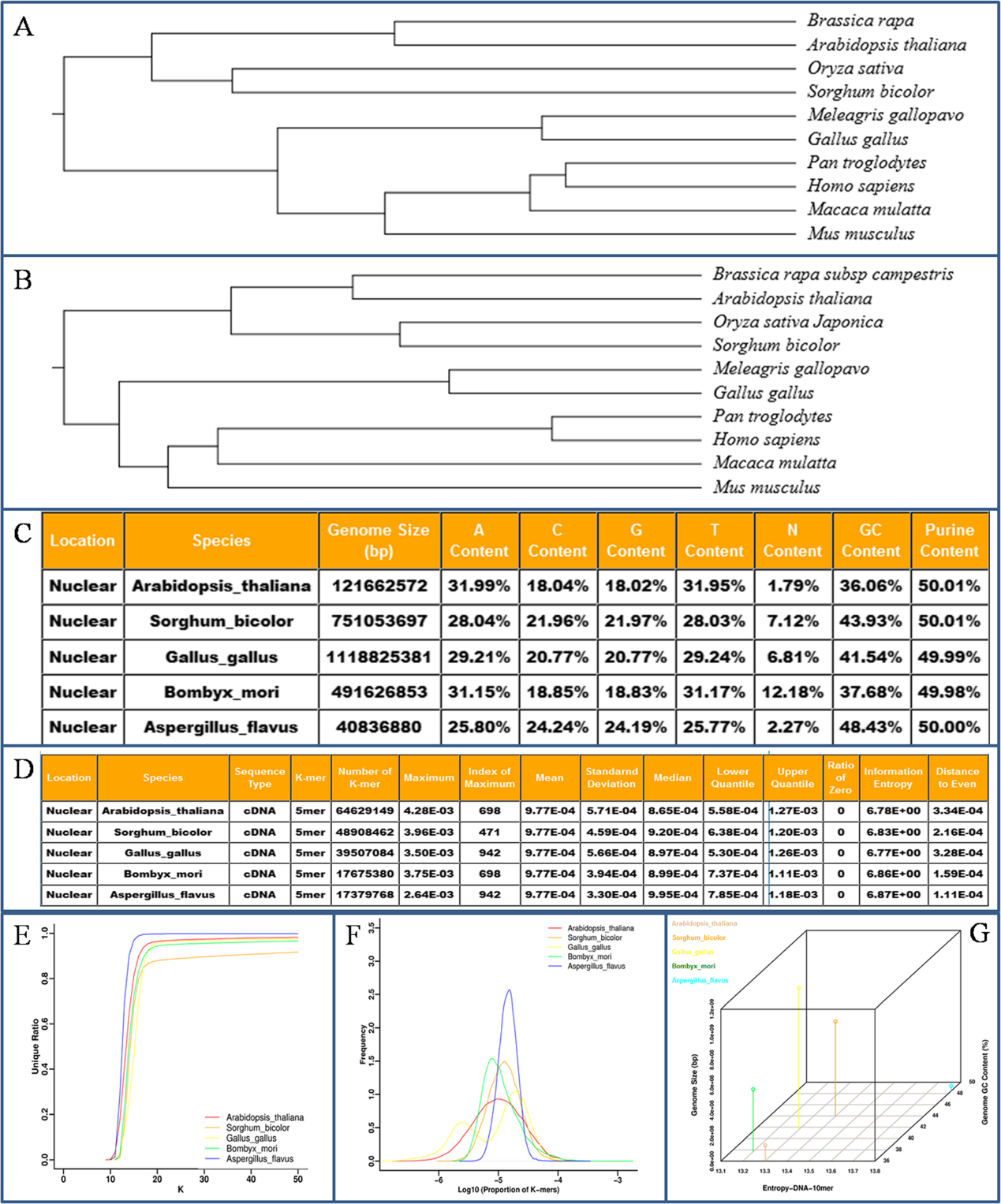
KGCAK數據庫中基本功能模塊舉例






