基因組所三代單分子測序PacBio完成技術升級—超長讀長助力基因組學研究
近日,基因組所所級中心基因組平臺三代單分子實時測序PacBio完成技術升級優化,實現了數據產量和讀長的雙重提升,一個SMRT Cell芯片可產出高達1Gb數據, reads平均長度達到14kb,N50超過19kb,為基因組學相關研究提供了有力支撐。
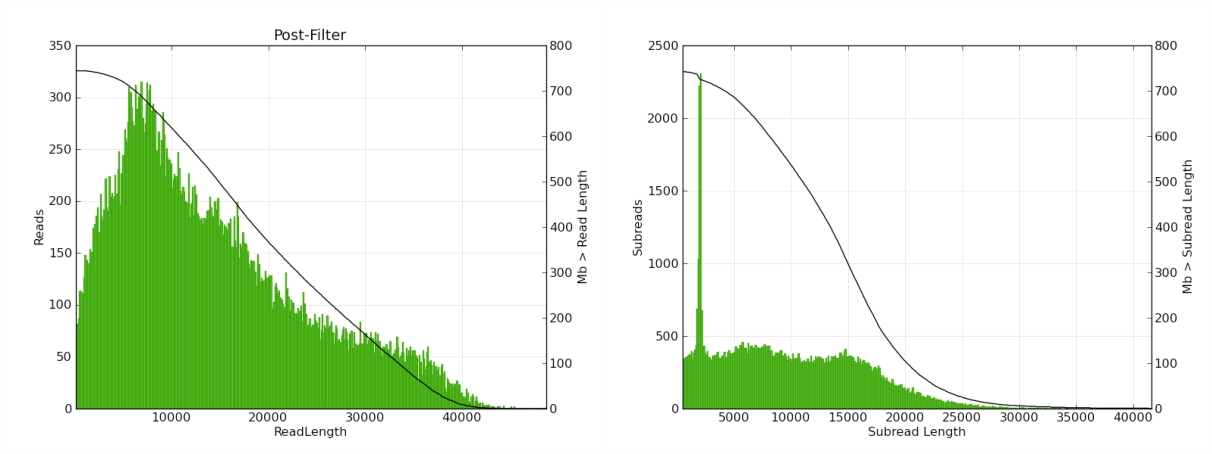
PacBio 測序read和subread長度分布
利用三代單分子實時測序儀PacBio系統,平臺提供的技術支撐服務包括:
1.基因組de novo測序組裝
PacBio測序read超長的特點,適合于基因組de novo組裝。例如一個5Mb大小的細菌基因組,只需一個SMRT Cell測序即可完成基因組finish,周期小于5天。對于復雜基因組,預期Contig N50超過100kb,推薦de novo組裝需50ⅹ以上的PacBio測序覆蓋,二代測序數據和PacBio測序數據混合組裝,推薦30ⅹ以上的PacBio測序覆蓋,如用于基因組scaffold構建,推薦10ⅹ以上的PacBio測序覆蓋。
2.全長轉錄本測序
受傳統測序技術短讀長的限制,較難獲得全長轉錄本序列。在PacBio測序平臺上,我們開發了全長cDNA測序和分析技術流程。PacBio測序產生的read長度遠大于絕大部分cDNA長度,因此無需經過后續序列拼接組裝,即可獲得準確全長cDNA序列,分析基因轉錄后可變剪切信息以及LncRNA表達等。
3.堿基修飾檢測
PacBio測序另一個優勢是可獲得DNA聚合酶促反應的分子動力學數據,分析堿基修飾信息。因為堿基基團修飾后的位阻作用,DNA聚合酶在帶有修飾堿基的核苷酸位置前后,表現出延滯效應,經計算分析得到IPD ratio差異,不同修飾的IPD ratio值不同,從而獲取堿基修飾信息。目前測序比較準確的修飾類型是m6A,另外5mC、5hmC、4mC等修飾信息也可得到,但需要特殊的樣品處理或數據處理。
此外,基于PacBio系統,平臺還提供基因組BAC克隆混合測序、長擴增子測序、極端GC含量DNA測序等技術開發服務。






