基因組所RNA m6A選擇性甲基化機制研究獲新進展
M6A(N6-methyladenosine,6-甲基腺嘌呤)是真核生物mRNA內部序列中最常見的一種甲基化修飾,同時受到甲基轉移酶(METTL3,METTL14,WTAP等)/去甲基化酶(FTO,ALKBH5等)以及一些RNA結合蛋白(YTHDF1/2/3, ELAVL1等)的共同調控,在細胞內是一個動態可逆的過程。敲除或抑制m6A相關的酶組分會導致重要的表型變化,如擬南芥胚胎滯育、人細胞凋亡、酵母配子發育缺陷,表明m6A可能是細胞內一種非常重要的修飾。
2012年,高通量測序與抗體免疫沉淀相結合方法(MeRIP-seq或m6A-seq)的提出,使得大規模檢測m6A修飾成為可能。近兩年,通過在人、小鼠、酵母和擬南芥等系統內的研究,在甲基化動態調控機制的方面取得了較大的進展,并發現m6A可能會直接或間接影響mRNA的出核轉運、翻譯和降解等。
近日,北京基因組所所級中心生物信息室宋述慧課題組,研究發現了RNA的選擇性甲基化現象,并探索了其可能的產生機制。該研究成果于2014年10月在RNA Biology在線發表。
科研人員采用m6A-seq技術進行了水稻愈傷與葉片兩個不同組織全轉錄組m6A的深度測序,并利用自主開發的軟件MeRIP-PF鑒定和繪制了水稻的首個m6A修飾譜,揭示了水稻m6A修飾譜的基本特征,即平均每個mRNA中有2~3個m6A修飾位點,并主要分布在CDS、3’UTR區域和起始密碼子區。
此外,在對數據的深度分析中,研究人員發現, m6A的甲基化存在組織或細胞的特異性和選擇性。研究人員將兩個組織(細胞系)中都表達但是只在某一個中發生甲基化的基因定義為選擇性甲基化基因(Selectively Methylated Genes, SMGs)。在測序的樣品中,分別鑒定到626個愈傷組織和5509個葉片組織的SMGs,通過對SMGs修飾峰的序列進行深入分析,探索了選擇性甲基化基因可能的產生機制,即某些RBP(如PUM蛋白)可能會作為甲基轉移酶結合mRNA時的“競爭者”,與甲基轉移酶競爭結合mRNA,從而產生組織或細胞系的SMGs。該項研究開啟了植物表觀轉錄組的研究,并提供了重要的數據資源。
該項研究工作得到了國家自然科學基金委,北京市科技新星的資助,同時還得到了精準基因組醫學重點實驗室楊運桂研究組和所級中心核酸平臺的技術支持。
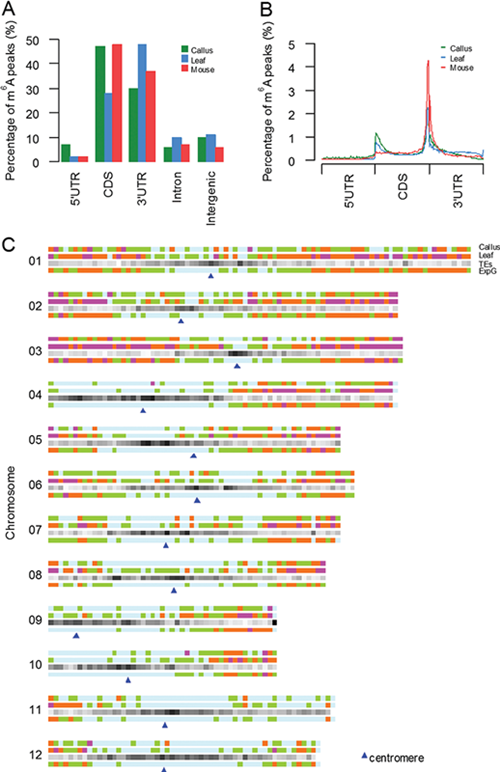
水稻愈傷和葉片組織RNA甲基化的全基因組分布圖






