基因組所開發“編碼蛋白質DNA序列并行比對工具—ParaAT”
中國科學院北京基因組研究所基因組科學與信息重點實驗室章張研究員,帶領其團隊成功開發“編碼蛋白質DNA序列并行比對工具—ParaAT(Parallel Alignment and back-Translation)”,該研究成果發表在Biochemical and Biophysical Research Communications雜志。
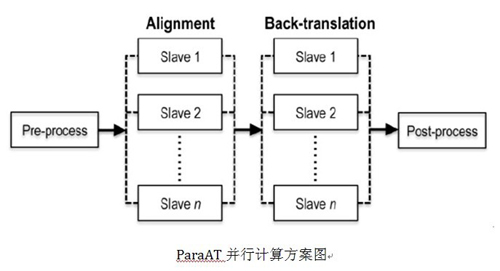
同源序列比對是生物信息學普遍采用的分析方法之一,其中,編碼蛋白質DNA序列比對最為常見,在比較基因組學、分子進化學、系統發育等領域具有重要的基礎作用。為獲取相應的比對結果,通常采用的方法是先比對后回譯(back-translate),此方法比直接進行DNA序列比對更可靠、準確。然而,現有相關的工具每次只能處理一組同源序列,無法實現多組同源序列的比對工作。
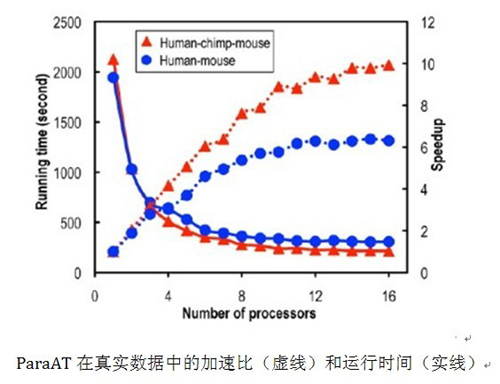
鑒于傳統工具所產生的弊端,基因組所科研人員采用并行計算(Parallel Computing)策略開發ParaAT,實現了多組同源編碼蛋白質DNA序列的并行比對。經驗證,ParaAT可大大降低運行時間,獲得較好的并行加速比(speedup),因此,適合大規模、同源序列的比對工作。
ParaAT可在不同操作系統下運行,支持多種不同的輸出格式,方便后續相關的生物信息學分析(諸如:用于檢測自然選擇壓力的KaKs_Calculator)。
文章鏈接:http://dx.doi.org/10.1016/j.bbrc.2012.02.101
軟件鏈接:http://cbb.big.ac.cn/software
附件下載:






