基因組所合作研究“水稻重要農藝性狀全基因組關聯分析和位點鑒定”獲突破
近日,中國科學院北京基因組研究所韓斌項目組利用新一代測序技術和自行開發的用于基因分型的算法,結合對水稻重要農藝性狀的群體遺傳學分析,開發了一套新的基因組關聯分析的技術和方法,實現了對控制水稻重要農藝性狀的候選基因的更加精確地篩選和鑒定。該項研究開創了新的基因組關聯分析的研究技術和方法,對復雜形狀相關基因的高效鑒定實現了新的突破。該研究論文已經被《自然*遺傳學》雜志接受發表。
水稻是世界上最重要的糧食作物之一,為世界上近一半的人口提供糧食。水稻在中國乃至全世界都有廣泛種植,在長期的水稻種植生產中,各地都保留了大量的水稻種質資源,具備很多優異的農藝性狀,是水稻育種改良的寶貴資源。如何高效鑒定全球栽培稻種質資源的遺傳多樣性以及快速、準確地挖掘水稻優良性狀相關基因,以更好地為水稻遺傳改良服務,是非常重要和具有挑戰性的研究課題。
韓斌項目組在上一年開展的中國水稻地方品種重測序和全基因組關聯分析的研究基礎上(論文發表在2010年Nature Genetics),又收集了來自世界各國的水稻種質資源,挑選了950份較有代表性的水稻品種,利用新一代基因組測序技術和自行開發的用于基因分型和缺失數據修復的算法,構建了一張精確的水稻高密度基因型圖譜,結合對水稻材料的群體遺傳學分析,該項目組準確鑒定了一些可能影響水稻群體分化的基因組區段和候選基因。通過對這950份水稻品種的農藝性狀進行了考察,包括抽穗期和十種籽粒性狀(包括產量、品質和顏色等三類性狀),我們在粳稻群體、秈稻群體和整個水稻群體中分別進行了全基因組關聯分析,鑒定到多個新的關聯位點。我們同時開發了一種基于單體型分析的局部基因組組裝方法,對基因區的不同等位基因分別進行組裝,鑒定序列變異。在定位到的關聯區域中,通過整合水稻基因注釋、芯片表達譜信息和序列變異信息,實現了更精確地對候選基因進行篩選和鑒定。
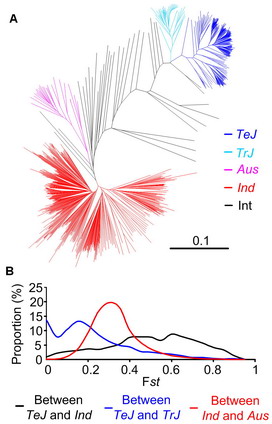
圖1:水稻材料的群體結構圖
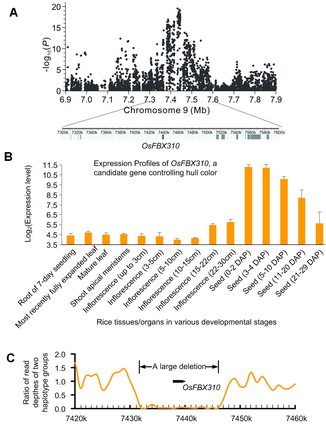
圖2:通過GWAS定位的控制水稻種殼顏色的候選基因及其表達模式和變異分析
Paper link: http://www.nature.com/ng/journal/vaop/ncurrent/full/ng.1018.html
Contact: Professor Bin Han Email: bhan@ncgr.ac.cn






